|

Kjetil Klepper
Avdelingsingeniør i bioinformatikk
|
Kontaktinformasjon:
- Kontor: 231.05.058 (Laboratoriesenteret, 5.etg Øst)
- Telefon: (+47)
72 57 33 61 .
- E-post: kjetil.klepper @ ntnu.no
- Postadresse:
Institutt for kreftforskning og molekylær medisin
Laboratoriesenteret, Erling Skjalgsons gt. 1
Det Medisinske Fakultet
Norges teknisk natur-vitenskapelige universitet (NTNU)
7006 Trondheim
|
|
|
Bakgrunn:
Ph.D., medisinsk teknologi (bioinformatikk), NTNU, 2013.
Cand. Scient., informatikk, NTNU, 2004. Fordypning i kunstig intelligens
Cand. Mag., informatikk, Universitetet i Oslo, 1999.
|
Forskning:
Et viktig satsningsområde for forskningsgruppa vår er å utvikle
gode og robuste metoder for å finne bindingsseter for transkripsjonsfaktorer
i DNA-sekvenser, eller å bedrive motivoppdaging som vi gjerne kaller det.
Hvis du aldri har hørt om transkripsjonsfaktorer eller motivoppdaging før,
foreslår jeg at du tar en titt på denne introduksjonen
som jeg har skrevet om emnet.
Mitt PhD-prosjekt: "Integrerte metoder for motivoppdaging i DNA"
Målet med prosjektet mitt er å utvikle et system som kombinerer forskjellige informasjonskilder
og metoder, med det formål å forbedre vår evne til å oppdage bindingsseter og motiver.
Et slikt system burde være i stand til å finne flere motiver enn tidligere metoder (økt sensitivitet),
samtidig som det unngår å foreslå motiver som ikke er reelle (økt spesifisitet).
Oppgaveformuleringen er således meget vid og åpner for mange interessante del-prosjekter.
Et sentralt element i systemet blir en modul for motivoppdaging basert på konsensus-prediksjon.
Dette er en teknikk fra kunstig intelligens som går ut på at man lar flere (i utgangspunktet
uavhengige) motivoppdagings- programmer predikere sett med motiver. Så kommer alle disse programmene sammen i en slags
jury hvor de stemmer over alle forslagene som er kommet inn. Majoriteten avgjør så i hvert tilfelle
om et forslag skal klassifiseres som et motiv eller ikke. Det fine med en slike metode er at den kan kombinere styrken
fra mange ulike programmer slik at resultatet til slutt blir bedre enn om man hadde brukt hvert program for seg selv.
Samtidig kan svake sider ved enkelte programmer tones ned og ikke få så store negative utslag.
Dersom et motiv er foreslått av flere ulike programmer er det nemlig mer sannsynlig at det her dreier seg om et ekte motiv,
mens motiver som er foreslått av kun ett program snarere er kandidater for å være falske positive.
Denne metoden bør dog ikke brukes ukritisk siden det godt kan hende at enkelte av programmene faktisk er mye bedre enn de andre og
finner motiver som ingen andre klarer å oppdage. Disse programmene burde selvfølgelig ikke straffes for å være flinke.
En oppgave blir derfor å finne gode måter å vekte stemmen til alle programmene i forskjellige situasjoner.
For eksempel kan det hende at noen programmer er bedre på visse typer datasett eller motiver,
og derfor burde deres stemme telle mer i disse tilfellene.
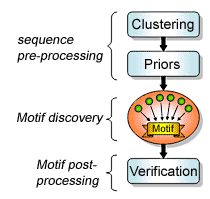
Det fullstendige systemet kan for eksempel følge den skjematiske fremstilligen i figuren til høyre,
hvor prosessen går gjennom tre trinn: pre-prosessering, motivoppdaging og post-prosessering.
Motivoppdagingsbiten vil hovedsakelig bestå av den nevnte modulen for konsensus-prediksjon.
Når så denne delen har foreslått et eller flere motiv, vil det neste steget naturlig være å undersøke
om disse motivene har noe for seg eller om de bare er tull. Et viktig steg i post-prosesseringen vil derfor
være å verifisere motiv-prediksjonene og eventuelt luke ut falske positive motiver som har sluppet igjennom.
En måte å gjøre dette på kan for eksempel være å undersøke om de foreslåtte motivene innehar visse egenskaper
som vi burde forvente å finne i ekte motiver. Hvilke egenskaper dette kan dreie seg om er foreløpig helt
ukjent, så et høyst aktuelt del-prosjekt vil nettopp være å avdekke om det i det hele tatt finnes slike
egenskaper som kjennetegner motiver og i så fall hva de kan være.
De fleste publiserte motivoppdagingsprogrammer forventer å bli fóret med et datasett hvor alle sekvensene antas
å inneholde de samme bindingsmotivene. Dersom mange av sekvensene ikke inneholder disse motivene, vil
det vanskeliggjøre motivoppdagingen siden forholdet mellom motiver og bakgrunn (eller "signal/støy-forholdet")
forskyves i negativ retning.
Det kan derfor være nyttig i en pre-prosesseringsfase å dele opp det opprinnelige datasettet i flere undergrupper
bestående av sekvenser med høyere likhet, og så gjøre motivoppdaging i hver av disse gruppene separat.
En annen faktor som er med på å redusere signal/støy-forholdet er at sekvensene i datasettet ofte kan være veldig
lange, mens selve bindingssetene bare utgjør en liten del. Hvis mulig kan det derfor være nyttig å veilede
motivoppdagingsprogrammene ved å tildele forskjellige deler av sekvensene ulike vekter som sier noe om
hvor sannsynlig det er at et bindingssete befinner seg i nettopp dette området av sekvensen. Teknikken kan
brukes til å for eksempel maskere ut repeterende elementer eller å tone ned områder som
med høy sannsynlighet er okkupert av nukleosomer og derfor ikke har aktive bindingsseter.
Sett fra et software engineering-perspektiv vil det være et mål å få til
et modulært system basert på en enkel og fleksibel arkitektur, slik at nye metoder og
teknikker som utvikles etterhvert bare kan plugges rett inn i systemet der hvor de hører hjemme.
|
|
|
Publikasjoner:
Klepper K and Drabløs F (2013) "MotifLab: a tools and data integration workbench for motif discovery and regulatory sequence analysis".
BMC Bioinformatics 14 : 9 [link] 
Fenstad MH, Johnson MP, Roten LT, Aas PA, Forsmo S, Klepper K, East CE, Abraham LJ, Blangero J, Brennecke SP, Austgulen R and Moses EK (2010) "Genetic and Molecular Functional Characterization of Variants within TNFSF13B, a Positional Candidate Preeclampsia Susceptibility Gene on 13q".
PLoS One 5(9) : e12993 [link]
Klepper K and Drabløs F (2010) "PriorsEditor: a tool for the creation and use of positional priors in motif discovery".
Bioinformatics 26(17) : 2195–7 [link]
Klepper K, Sandve G, Abul O, Johansen J and Drabløs F (2008) "Assessment of composite motif discovery methods".
BMC Bioinformatics 9 : 123 [link] 
|
Annet:
Her kan du finne min personlige hjemmeside.
Og her er en liste over alle spennende artikler som jeg foreløpig
har lest i forbindelse med doktorgrads-studiene.
Her er alle fagene jeg har tatt som student.
|
|
|
|
|
|